第一章 引言
一、冠状病毒结构及分类
冠状病毒(coronavirus,CoV)属于冠状病毒科、尼多病毒目的成员之一,是广泛存在于自然界的一大类病毒,可感染人及多种动物[1, 2],并且对人及家畜具有巨大的潜在威胁。冠状病毒的宿主范围广泛,目前已在人及骆驼、猪、牛、鸡、鸭、鹅、猫、狗、鼠和蝙蝠等动物中分离到 CoV,可引起呼吸道、消化道和神经系统等的感染[3]。
CoV 因其在电子显微镜下为典型的冠状糖蛋白而得名,其大小在 27kb-32kb之间,是目前已知最大的单链正义 RNA 基因组[3]。病毒基因组的 3’端具有 poly(A)尾,5’端具有甲基化的帽状结构[4]。通常,冠状病毒三分之二的基因组 RNA 编码两个大的重叠多蛋白 ORF1a 和 ORF1ab,它们被加工成病毒聚合酶(RdRp)和参与 RNA 合成或宿主反应调节的其他非结构蛋白。而基因组的另外三分之一编码四种结构蛋白(刺突(S),包膜(E),膜(M)和核衣壳(N))和其他辅助蛋白。
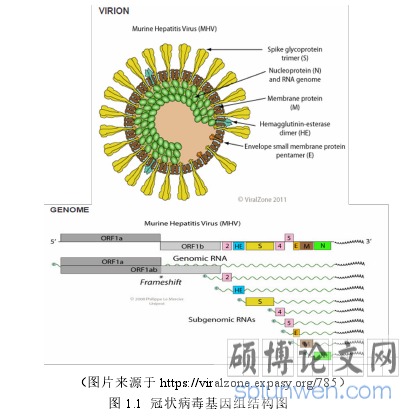
.........................
二、冠状病毒感染细胞过程
动物病毒的增殖过程一般分为五个阶段:吸附、穿入、脱壳、病毒组分的合成、装配与释放。它是一种必须在活细胞内寄生并以复制方式增殖的非细胞型微生物,因此,进入细胞是其完成复制周期的首要条件,而又因病毒颗粒很大,不能通过自由扩散穿过细胞膜,病毒需要首先粘附至细胞膜上,此环节由病毒与易感细胞表面的特异性结合受体相互结合而完成[1, 24]。

...........................
第二章 高致病性冠状病毒密码子偏爱性分析
一、冠状病毒结构及分类
冠状病毒(coronavirus,CoV)属于冠状病毒科、尼多病毒目的成员之一,是广泛存在于自然界的一大类病毒,可感染人及多种动物[1, 2],并且对人及家畜具有巨大的潜在威胁。冠状病毒的宿主范围广泛,目前已在人及骆驼、猪、牛、鸡、鸭、鹅、猫、狗、鼠和蝙蝠等动物中分离到 CoV,可引起呼吸道、消化道和神经系统等的感染[3]。
CoV 因其在电子显微镜下为典型的冠状糖蛋白而得名,其大小在 27kb-32kb之间,是目前已知最大的单链正义 RNA 基因组[3]。病毒基因组的 3’端具有 poly(A)尾,5’端具有甲基化的帽状结构[4]。通常,冠状病毒三分之二的基因组 RNA 编码两个大的重叠多蛋白 ORF1a 和 ORF1ab,它们被加工成病毒聚合酶(RdRp)和参与 RNA 合成或宿主反应调节的其他非结构蛋白。而基因组的另外三分之一编码四种结构蛋白(刺突(S),包膜(E),膜(M)和核衣壳(N))和其他辅助蛋白。

.........................
二、冠状病毒感染细胞过程
动物病毒的增殖过程一般分为五个阶段:吸附、穿入、脱壳、病毒组分的合成、装配与释放。它是一种必须在活细胞内寄生并以复制方式增殖的非细胞型微生物,因此,进入细胞是其完成复制周期的首要条件,而又因病毒颗粒很大,不能通过自由扩散穿过细胞膜,病毒需要首先粘附至细胞膜上,此环节由病毒与易感细胞表面的特异性结合受体相互结合而完成[1, 24]。
冠状病毒复制周期是从翻译病毒基因组的5’端ORF开始(ORF1a和ORF1ab),其合成两个多聚蛋白 pp1a 和 pp1ab[25]。通过 ORF1a 编码的蛋白酶进行酶切后,从pp1a 和 pp1ab 上释放 15 或 16 个复制蛋白酶,在复制酶的作用下,病毒基因组通过不连续转录模式(Discontinuous transcription mode),转录生成新的基因组 RNA和 mRNA,接着进入细胞质,进行翻译工作:基因组 RNA 首先开始翻译,产生病毒 RNA 复制所需的蛋白酶;其次是亚基因组翻译,产生 S、M、N 和 E 等结构蛋白。在病毒组装过程中,其结构蛋白 N 与病毒结合,形成复合体形态,该复合体可以通过结构蛋白 M 蛋白、E 蛋白的相互作用,迅速被包裹到核衣壳中。随后在出芽的过程中,S 蛋白整合到病毒中,形成冠状结构。最后成熟的病毒颗粒在 M蛋白的作用下从囊泡中释放出来[26]。具体过程见图 1.2(以 SARS-CoV、MERS-CoV为例):

...........................
第二章 高致病性冠状病毒密码子偏爱性分析
第一节 六种人冠状病毒密码子偏爱性比较
1 实验材料
本研究涉及的冠状病毒基因编码序列(CDS)均来自 NCBI 数据库(https://www.ncbi.nlm.nih.gov/)。
2 实验方法
本研究使用 EMBOSS(http://emboss.toulouse.inra.fr/)子程序 CUSP 计算密码子 Frequency 值[78],CodonW(https://sourceforge.net/projects/codonw/)计算密码子ENC、GC、GC3S、RSCU 值[79],Lasergene 子程序 EditSeq 保存截取的蛋白编码序列。使用 Sigmaplot 绘图、SPSS22.0 进行聚类分析。
3 实验结果
3.1 有效密码子数目分析
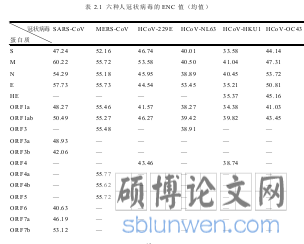
..........................
.........................
1 实验材料
本研究涉及的冠状病毒基因编码序列(CDS)均来自 NCBI 数据库(https://www.ncbi.nlm.nih.gov/)。
2 实验方法
本研究使用 EMBOSS(http://emboss.toulouse.inra.fr/)子程序 CUSP 计算密码子 Frequency 值[78],CodonW(https://sourceforge.net/projects/codonw/)计算密码子ENC、GC、GC3S、RSCU 值[79],Lasergene 子程序 EditSeq 保存截取的蛋白编码序列。使用 Sigmaplot 绘图、SPSS22.0 进行聚类分析。
3 实验结果
3.1 有效密码子数目分析

..........................
第二节 SARS-CoV 与 SARSr-Co V 密码子偏爱性比较及聚类分析
1 实验材料
本研究涉及的冠状病毒基因编码序列(CDS)均来自 NCBI 数据库(https://www.ncbi.nlm.nih.gov/)。
2 实验方法
方法同上节 2。
3 实验结果
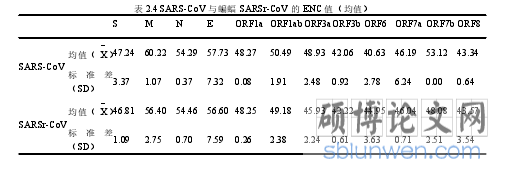
3.1 有效密码子数目分析
SARS-CoV 的全基因组由 29727 个核苷酸组成,由 11 个开放阅读框(ORF)。共编码 S、E、M 和 N 4 种结构蛋白和 ORF1a、ORF1b、ORF3a、ORF3b、ORF6、ORF7a、ORF7b、ORF8 等多种功能未知的蛋白[86]。研究表明,SARS-CoV 与SARSr-CoV 各蛋白有效密码子数目(ENC)值在 40-58 之间,均接近 61,并且,两者相同蛋白的 ENC 值非常接近(如表 2.4 所示)。
1 实验材料
本研究涉及的冠状病毒基因编码序列(CDS)均来自 NCBI 数据库(https://www.ncbi.nlm.nih.gov/)。
2 实验方法
方法同上节 2。
3 实验结果
3.1 有效密码子数目分析
SARS-CoV 的全基因组由 29727 个核苷酸组成,由 11 个开放阅读框(ORF)。共编码 S、E、M 和 N 4 种结构蛋白和 ORF1a、ORF1b、ORF3a、ORF3b、ORF6、ORF7a、ORF7b、ORF8 等多种功能未知的蛋白[86]。研究表明,SARS-CoV 与SARSr-CoV 各蛋白有效密码子数目(ENC)值在 40-58 之间,均接近 61,并且,两者相同蛋白的 ENC 值非常接近(如表 2.4 所示)。

.........................
第三章 高致病性冠状病毒 S 蛋白的真核表达载体的构建...................35
第一节 SARS-CoV S 蛋白真核表达载体的构建.......................................36
1 实验材料.................................36
2 实验方法...........................37
第四章 全文小结...............................55
第三章 高致病性冠状病毒 S 蛋白的真核表达载体的构建
第一节 SARS-CoV S 蛋白真核表达载体的构建
1 实验材料
1.1 菌株 、质粒
大肠埃希菌(DH-5α)甘油菌购自天根生化科技有限公司;重组 DNA 克隆载体 pcDNA3.1 为本实验室保存。真核表达载体 pcDNA3.1,大小为 5428bp。pcDNA3.1-SARS 为本实验室保存。
将称好的 242 g Tris 碱、37.2 g Na2 EDTA·H2O;搅拌溶解,加入 57.1 mL 冰乙酸;调 PH 至 8.3;充分搅拌后定容至 1000 mL,
4℃保存。
将称好的 10 g 胰蛋白胨、5 g 酵母提取物和 10 g Na Cl 溶解于适量去离子水中,用磁力搅拌器搅拌均匀后定容至 1 L,分装于试管中,然后高温高压灭菌 30 min,降至室温后,4 ℃保存。
将称好的 4 g 琼脂,加入到 200 mL 液体 LB 培养基中,于高温高压下灭菌 30min,在未凝固前,工作超净台下,以无菌操作注入无菌培养皿中,室温凝固后,4 ℃保存。
............................
第四章 全文小结
1. 本文采用分子生物信息学技术,分析比较了高致病性冠状病毒与普通 HCoV、不同宿主间高致病性冠状病毒的密码子偏性,发现它们在密码子的选择上具有极大的相似性。
2. 本文从密码子偏性角度基于六种 HCoV、不同宿主间高致病性冠状病毒的单个基因进行聚类分析,发现六种 HCoV 基于密码子偏性分析的聚类分析与基于它们全基因组的进化树分析结果不同;云南蝙蝠 SARSr-CoV 可能是 SARS-CoV、中国其他省份 及行 政区蝙蝠 SARSr-CoV 的 自然疫源地 ;骆驼 MERS-CoV 与人MERS-CoV 在近三年交流比较频繁,这进一步从密码子偏性角度证实了 WHO 的最新观点。
3. 本文利用重叠 PCR 技术克隆 SARS-CoV、MERS-CoV 的 S、S1、RBD 基因编码序列,成功构建相关真核表达载体。
参考文献(略)
............................
第四章 全文小结
1. 本文采用分子生物信息学技术,分析比较了高致病性冠状病毒与普通 HCoV、不同宿主间高致病性冠状病毒的密码子偏性,发现它们在密码子的选择上具有极大的相似性。
2. 本文从密码子偏性角度基于六种 HCoV、不同宿主间高致病性冠状病毒的单个基因进行聚类分析,发现六种 HCoV 基于密码子偏性分析的聚类分析与基于它们全基因组的进化树分析结果不同;云南蝙蝠 SARSr-CoV 可能是 SARS-CoV、中国其他省份 及行 政区蝙蝠 SARSr-CoV 的 自然疫源地 ;骆驼 MERS-CoV 与人MERS-CoV 在近三年交流比较频繁,这进一步从密码子偏性角度证实了 WHO 的最新观点。
3. 本文利用重叠 PCR 技术克隆 SARS-CoV、MERS-CoV 的 S、S1、RBD 基因编码序列,成功构建相关真核表达载体。
参考文献(略)
