本文是一篇软件工程论文,本研究在多序列比对方面提出的MitoAlign算法有效解决了传统手工比对效率低下、精度欠佳的难题,极大地推动了相关领域研究的进展,为后续深入探究线粒体基因组特征奠定了基础。
1 绪论
1.1 研究背景及意义
自1953年沃森和克里克发现DNA双螺旋结构后,分子生物学进入快速发展阶段。分子生物学作为在分子层面解析生命现象的学科,核心研究内容包括蛋白质、DNA和RNA等生物大分子的结构与功能。随着科学研究的不断深入,尤其是在基因组学、转录组学等领域的突破性进展,逐渐积累了海量的生物数据。随着这些数据量的激增,传统的实验手段和观察方法已难以满足现代研究的需求,因而逐渐依赖于计算机科学、数学等学科的技术来揭示海量数据背后隐藏的规律,这种跨学科的结合就导致了一门新兴的交叉学科——生物信息学。生物信息学是以生物大分子数据集为研究对象,研究理论模型和方法,旨在揭示基因组、转录组和蛋白质组的生物复杂性,以及生命现象的根本规律,例如生长、发育、遗传、进化和疾病[1–4]。序列相似性分析和序列比对是计算分子生物学的基础步骤[5–7]。
为了研究一个未知基因的功能,生物学家通常采用将其与已知基因作比较的方法,并通过数据库检索获取与其相似的基因来进行分析。这种方法的关键在于序列比对技术,目前最常用的包括FASTA[8]和BLAST(Basic Local Alignment Search Tool)[9]等技术。1983年,一项有趣的研究揭示了这种方法的重要性[10]。当时,科学家发现了一个新的致癌基因v-sis,并将其与已知基因进行对比。经过比对,研究者发现该致癌基因与血小板生长因子基因(platelet-derived growth factor,PDGF)高度相关。这个发现促使科学家们开始思考:癌症是否由某些正常的生长基因在演变过程中发生突变引发。如今,多序列比对技术已经被广泛应用,为生物基因数据的分析提供了快速和精确的解决方案,也为基因组学的研究开辟了新的技术路径。
软件工程论文怎么写
..............................
1.2 国内外研究现状
本章围绕线粒体基因组研究的两个核心方向——多序列比对与基因重排量化展开,系统梳理了国内外研究进展与技术突破。
1.2.1 多序列比对研究
多序列比对是一个复杂的问题,因此目前的多序列比对方法通常将其简化为多个双序列比对(Pairwise Sequence Alignment,PSA)。双序列比对通常将一条待比对的查询序列与参考序列进行比对,最常见的应用场景是在完成测序后,为每个测序序列找到其在参考基因组中的准确比对位置。相比之下,双序列比对较为简单,其核心在于寻找最优比对,即根据给定的目标函数,确定序列中核苷酸或氨基酸片段的最佳对齐,这通常可以用动态规划算法直接解决。动态规划本质上依赖最优原则,被视为解决某些组合优化问题的有效方法,因此在生物信息学中具有重要应用,特别是在核苷酸序列和氨基酸序列的比对中作为基础方法[24]。
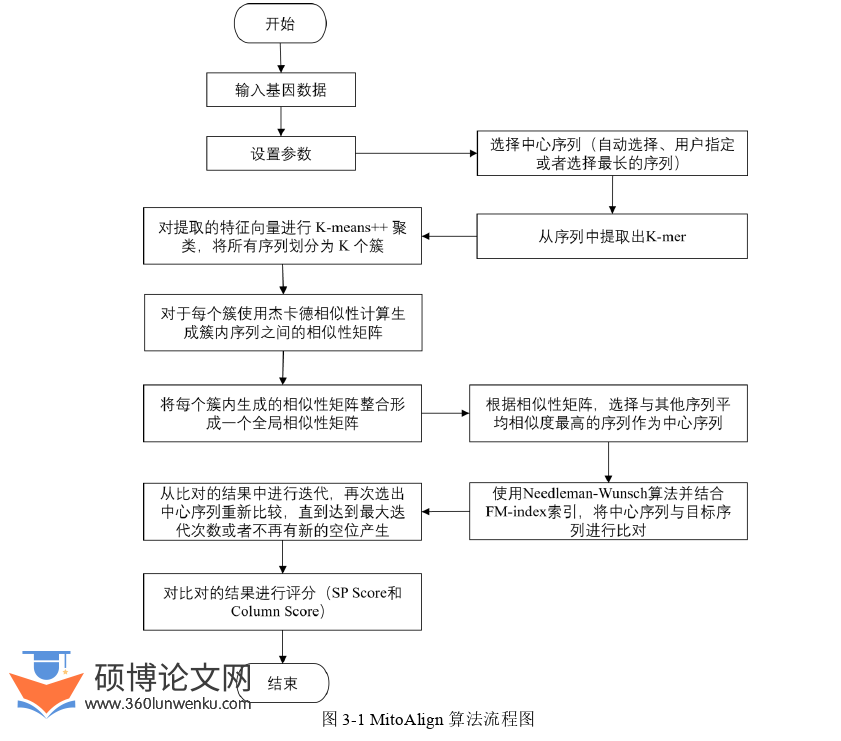
双序列比对的空间复杂度为O(n²)(n为两条序列的平均长度),因此多序列比对就变成了一个时间复杂度为O(nm)(m为序列数量)的高维问题。由于存在多条路径组合,使得多序列比对成为一个NP-hard问题[25]。因此,在多序列比对中,精确比对所需的计算量巨大且耗时较长,直接采用动态规划处理多条序列在实际应用中不可行。所以生物计算中通常不再追求精确的最优解,而是通过估计获得一个近似解[26]。具体方法是将多序列比对转化为一系列双序列比对,整个过程通常包括三个关键步骤:首先计算所有序列对之间的相似性;其次根据相似性组织序列,通常通过构建指导树或选择最相似的中心序列;最后根据指导树或中心序列逐步完成双序列比对,从而得到整个序列集的多序列比对结果。
.............................
2 相关概念和方法
2.1 线粒体基因数据
线粒体基因组作为真核生物细胞器基因组的典型代表,其结构特征与功能组织呈现高度保守性与特化性的统一。该基因组通常以15~20 kb的闭环分子形式存在,编码37个基因单元,包括13个呼吸链相关蛋白编码基因(如 ND1-6、COX1-3 等)、2个核糖体 RNA(12S rRNA 与 16S rRNA)及22个转运 RNA 基因。这些基因产物通过模块化组装参与氧化磷酸化系统的构建,其中蛋白质编码基因的产物分别定位于呼吸链复合体 I(NADH脱氢酶)、III(细胞色素 bc1 复合体)、IV(细胞色素c氧化酶)及V(ATP合酶)。值得注意的是,大多数昆虫线粒体基因呈现显著的链偏向性分布,约90%的基因位于J链(重链),剩余基因(如nad6及8个tRNA基因)则分布于N链(轻链),这种不对称分布模式与转录调控及复制起始密切相关。
线粒体基因组的非编码区域具有重要的调控功能,其中控制区(control region)包含关键的复制起始位点与转录调控元件。该区域在昆虫与线虫中表现出显著的 AT 碱基偏好性(可达80%以上),故被称为 AT 富集区。以人类线粒体基因组为例,其控制区(D-loop 区)包含保守序列块(CSB)和终止结合序列(TAS),通过形成特定的二级结构调控 DNA 复制与转录起始。值得强调的是,线粒体基因组的紧凑性特征尤为突出,基因间间隔区通常小于50 bp,部分区域甚至存在基因重叠现象(如ATP8与ATP6基因存在40 bp的重叠序列),这种高密度的基因排列方式与其能量代谢的高效性需求高度契合。
......................
2.2 多序列比对算法的相关概念和方法
2.2.1 基本概念
1. K-mer片段
生物序列属于大规模序列数据,其中往往存在大量相同或相似的片段。在进行序列相似性搜索时,索引技术能够高效地组织庞大的序列数据,从而提高数据的查询效率。因此,索引技术在生物序列分析中,如序列比对、序列分组等工作中,一直发挥着重要作用。在本节中,将重点介绍K-mer片段。
K-mer是指长序列中长度为q的短序列。对序列中所有的K-mer进行索引,在相似序列搜索时,可通过定位序列间公共的K-mer有效缩小搜索范围。获取 K-mer的方法较为简单,将长度为q的窗口从序列起始位置开始,每次向右滑动m个字符,直至窗口到达序列的结束位置。当m的值为1时,便能获取一条序列上的所有 K-mer。K-mer索引通过连续存储每个K-mer出现的所有位置,查询一个K-mer即可得到其出现的所有位置,每个K-mer会对应一个哈希值。因此,K-mer索引实际上是基于哈希索引特性实现的,采用两个表来存储数据:一个是 K-mer表,另一个是地址表。地址表记录了所有K-mer在序列上出现的位置,每个K-mer在序列上出现的位置被连续记录,并按照K-mer哈希值大小排序。K-mer 表的地址对应序列中所有不重复 K-mer 的哈希值,每个地址记录了相应 K-mer 在地址表中首次出现的偏移位置。
.....................
3 融合中心星和迭代的线粒体基因组多序列比对算法 ............................. 19
3.1 算法设计思想 ............................ 19
3.1.1 数据预处理与参数初始化 ................... 20
3.1.2 核心算法执行框架 .......................... 21
4 基于区域加权的线粒体基因组重排量化算法 .................... 36
4.1 算法设计 ........................................ 36
4.2 实验结果与分析 ........................... 39
4.3 原型工具开发 ............................ 44
5 总结与展望 ................................ 47
5.1 总结 ................................. 47
5.2 展望 ............................. 48
4 基于区域加权的线粒体基因组重排量化算法
4.1 算法设计
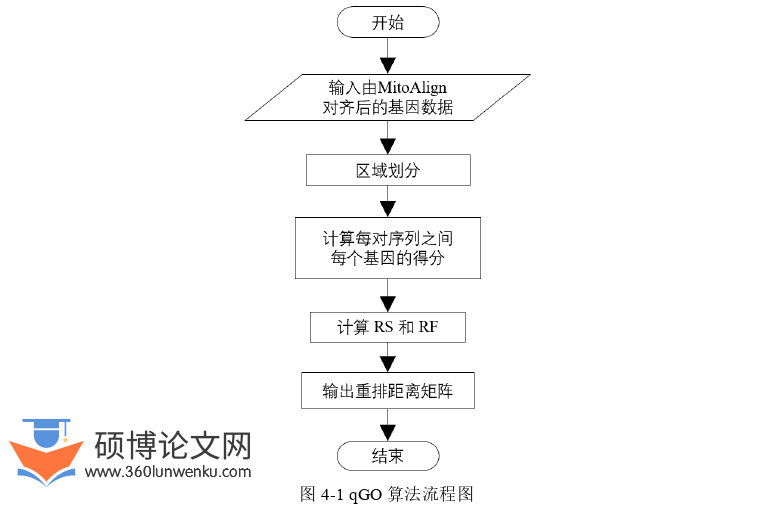
基因组是染色体的集合,每个染色体代表一个基因序列。基因在染色体中的方位是通过在基因串前添加符号±(加号可以省略)来表示的。例如,{(F, -12S, W, A, -N)} 表示包含五个基因的单染色体基因组[60]。基因组可以是线型的,也可以是环型的。在环状基因组中,序列的最后一个基因与第一个基因相连,并排列成环状。另一方面,基因是线性排列的,有头有尾。在本研究中,我们只考虑单染色体环状基因组。包含m个(m≥0)基因的染色体Si (0 ≤ i ≤ n-1)表示为 {g0 g1 … gm-1},其中 gj(0≤j≤m-1)。
NCBI数据库中后生动物发生重排的线粒体基因组可分为以下3种主要重排类型:(1)滑移(shuffling):基因在同一条链上从原来位置移到相邻位置(一般不跨越蛋白基因);(2)移位(translocation):基因从原来位置跨过几个基因(通常包括蛋白质编码基因)移位到不同的位置;(3)倒置(inversion):基因从某一条链编码转换到另外一条链编码[61]。通过比较分析NCBI目前已公布的26种异尾次目线粒体基因组排序后,可以发现该类群线粒体基因组均发生了大规模的基因重排,并且只涉及移位和倒置2种重排类型[62]。本研究仅对这2种重排类型进行分析。
软件工程论文参考
.........................
5 总结与展望
5.1 总结
多序列比对与基因重排量化作为生物信息学的重要课题,在构建系统发育树、基因功能注释及进化分析等领域具有重要应用价值。本文的主要工作是针对线粒体基因组的多序列比对和基因重排量化问题。首先,本文提出了MitoAlign算法,该算法创新性地将中心星比对算法和迭代比对算法相结合,发挥了它们各自的优势,并进行了一系列的优化,包括改进k-mer特征提取方法、优化K-means++聚类和快速构建杰卡德相似性矩阵。在降低比对时间的同时,也维持了较高的比对精度。其次,开发基于基因顺序同源性的新型统计方法——qGO,通过区域加权策略实现线粒体基因组多样性量化。总结如下:
(1)提出了一种新型线粒体基因组多序列比对算法MitoAlign。该算法采用分治策略,基于K-mer特征的K-means++聚类将序列划分为子集,结合杰卡德相似度矩阵、中心星和迭代优化算法,实现线粒体基因组的多序列比对。
(2)在K-mer特征提取过程中,通过并行化架构提升大规模数据处理效率。采用哈希映射和位数组存储实现去重优化,降低内存占用。并构建双层索引结构:首先生成全局唯一K-mer字典,采用哈希算法生成64位标识符确保跨物种比对一致性;继而构建物种K-mer的二进制关联矩阵,通过双哈希排序策略(字典序+物种特异性哈希)实现快速定位。
(3)提出改进的K-means++初始化策略,基于概率选择机制优化初始质心分布,在保证算法收敛性的同时提升聚类稳定性。该概率模型通过提升远距离样本的选择概率,避免质心聚集,实现初始质心的均匀分布,有效降低随机初始化引发的局部最优风险。同时引入DBI作为聚类质量的验证指标。该指数通过量化聚类簇的紧凑性与分离性,实现聚类质量的综合评估。
参考文献(略)
